EC-isHCR: a rapid method for in situ hybridization chain reaction in diverse animal samples (EC-isHCR:多様な動物サンプルに適用可能な高速 in situ ハイブリダイゼーション連鎖反応法)
Yasuhiro Kozono, Satoru Kobayashi et al. (Degree Programs in Life and Earth Sciences, Graduate School of Science and Technology, University of Tsukuba, Japan)
論文要旨
in situハイブリダイゼーション連鎖反応(isHCR)は、多様な試料において臓器から細胞内構造に至る複数の空間スケールにわたるRNAを可視化する。我々は以前、ショウジョウバエ胚および卵巣向けの迅速プロトコルEC-isHCRを提案した。しかし、EC-isHCRが従来のisHCRの特徴(様々な試料における広範囲な空間スケール解析を含む)を維持しているかどうかは不明であった。本研究では、EC-isHCRが果蠅の全標本、寄生蜂、アブラムシの標本、マス(トラウト)のパラフィン切片、マウスの凍結切片、ヒト培養細胞など、幅広いサンプルにおいて堅牢なRNA検出を可能にすることを示した。さらにEC-isHCRは細胞内RNA局在の検出を可能とし、ショウジョウバエ胚における相分離凝縮体との共局在を可視化、HeLa細胞における突起部濃縮mRNAの検出にも成功した。EC-isHCRの適用範囲拡大のため、自動プローブ設計ツール(https://github.com/ShuntaYorimoto/hcrkit)を開発した。このツールとEC-isHCRを組み合わせることで、mRNA可視化のための迅速かつ汎用性の高いフレームワークを提供する。本フレームワークは高速isHCR利用の障壁を低減し、生命科学の多様な分野における研究促進に寄与する。
Abstract
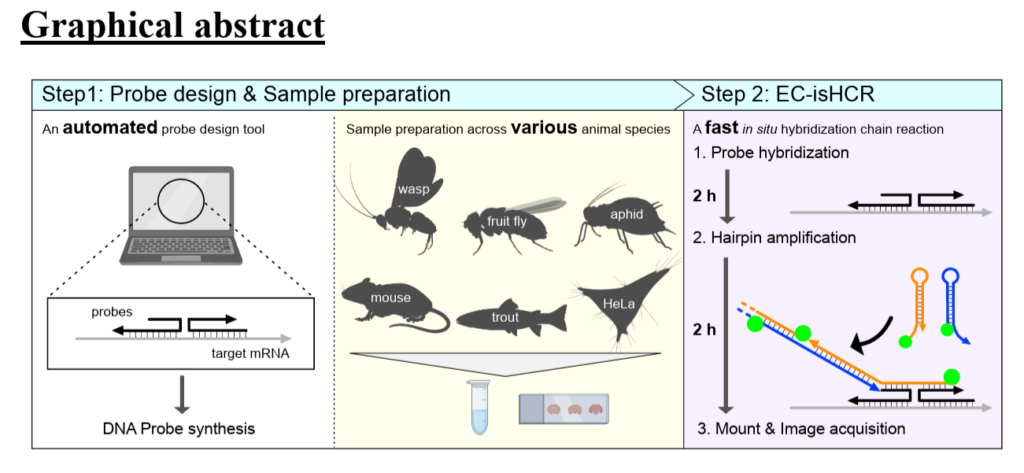
本研究は、in situ hybridization chain reaction(isHCR)を高速化した手法であるEC-isHCRの汎用性を検証した。EC-isHCRは、ショウジョウバエ、寄生蜂、アブラムシの全胚・全個体、ニジマスのパラフィン切片、マウス凍結脳切片、ヒト培養細胞など、幅広いサンプルにおいて高コントラストで安定したRNA検出を可能にした。さらに、サブセルラー局在(RNA顆粒や細胞突起)も可視化できた。加えて、EC-isHCR用の自動プローブ設計ツール(hcrkit)を開発し、迅速かつ汎用的なmRNA可視化フレームワークを提示した。
Background
従来の第3世代isHCRや改変isHCRは高感度かつ多様なサンプルに適用可能である一方、染色に全胚で約3日、切片で約2日を要するという欠点があった。著者らは以前、エチレンカーボネート(EC)を用いることで反応時間を短縮したEC-isHCRをショウジョウバエ胚で報告したが、その汎用性や空間解像度は未検証であった。
Methods
- EC-isHCR:15%エチレンカーボネートを含むハイブリダイゼーションバッファーを用い、プローブハイブリダイゼーション2時間(45℃)、ヘアピン増幅2時間(25℃)で完了。
- 比較実験:ニジマス精巣切片で改変isHCR(2日)とシグナル強度を比較。
- サンプル:ショウジョウバエ、D. sechellia、寄生蜂(Asobara japonica)、アブラムシ(Acyrthosiphon pisum)、ニジマス、マウス、HeLa細胞。
- プローブ設計:GC含量40–60%、BLASTによるオフターゲット評価を自動化した新ツール(hcrkit)を使用。
Results
- 多様な動物種・試料での適用性:
- ショウジョウバエの生殖腺・腸・副生殖腺でvasa、tj、esg、Tk、SP mRNAを検出。
- 寄生蜂の毒腺でIDDF-1 mRNA、アブラムシ胚でBCR1およびvasa1 mRNAを検出。
- ニジマス精巣切片でvasa(生殖細胞)とgsdf(セルトリ細胞)を明瞭に検出。
- マウス脳切片でGad1(RF高発現)とPenk(線条体特異的)を検出。
- 比較:EC-isHCRは改変isHCRと同等のシグナル強度を示し、所要時間は1日に短縮。
- 細胞内局在:
- ショウジョウバエ胚の生殖顆粒におけるnanos mRNA局在。
- HeLa細胞突起へのKIF1C mRNA集積。
Discussion
この論文のDiscussionは、「EC-isHCRは速いだけでなく、従来isHCRの強み(多様なサンプル適用・多階層スケール・高感度)を維持している」ことを、実験結果と既存法との比較を通して整理し、さらにこの枠組み(EC-isHCR+自動プローブ設計ツール)が研究現場で何を変えるかを論じている。
1. 適用範囲が広く、従来の利点を保ったまま高速化できた
著者らは本研究で、EC-isHCRが以下のように幅広いサンプルでmRNA検出できることを示した。
- 全載標本:ショウジョウバエ、寄生蜂、アブラムシ
- 切片:ニジマス(パラフィン)、マウス(凍結)
- 培養細胞:ヒト(HeLa)
この点をもって、EC-isHCRは固定法や試料形態が異なるサンプルに対応できる可能性が高いと議論している(=汎用性の主張)。
2. 「多階層スケールで見える」=従来isHCRの核となる特徴を維持
Discussionの重要点は、EC-isHCRが単に「染まる」だけではなく、従来isHCRが得意とする複数の空間スケールをカバーできたことです。著者らは、次の3階層を明示して整理しています。
- 臓器レベル:ショウジョウバエ雄副生殖腺、寄生蜂毒腺、マウス脳
- 細胞レベル:ショウジョウバエ幼虫生殖腺・腸、アブラムシのバクテリオサイト、ニジマス精巣
- サブセルラーレベル:ショウジョウバエ胚の生殖顆粒、HeLa細胞の突起
ここから著者らは、EC-isHCRは従来isHCRの「多スケール解析が可能」という本質的価値を保っている、と結論づけている。
3. 感度・シグナル強度を落としていない(=高速化の代償が小さい)
次に著者らは、「速いけど弱い」のでは困るので、シグナル強度(≒検出感度)を維持できている根拠を論じる。
- ニジマス切片で、EC-isHCRのシグナル強度がmodified isHCRと同等だった(Fig.3)
- さらに、過去の報告で第3世代isHCRやmodified isHCRで検出されていたmRNAを、EC-isHCRでも検出できた(複数遺伝子で整合)
これらから著者らは、EC-isHCRは感度を犠牲にせずに時間短縮できる可能性が高い、と位置づけている。
4. プローブ設計ツールの改良が「普及のボトルネック」を解消する
Discussionの後半で大きいのが、実験手法そのものだけでなく、使うまでの障壁(プローブ設計)を下げた点の議論である。
著者らは「以前もプローブ設計ツールを公開していたが、制約があった」とし、具体的に:
- BLASTによるオフターゲット評価が手動で、使いづらい
- 候補領域の重なり排除を先にやる設計だと、BLASTで合格する候補が落ちてしまい、最終的なプローブ数が減る
そこで新ツールでは、
- ローカルBLASTをワークフローに統合して自動オフターゲット評価
- 重なりを許して候補抽出→BLAST評価→最後に非重複選択に変更し、採用可能な候補を増やす
という改善を行い、EC-isHCRの適用範囲をさらに広げた、と説明している。
5. 他法(RNAscope / smFISH)との比較
著者らは「現場での選択肢としてどうか」を示すため、代表的ISH法と比較している。
RNAscopeとの比較(主に“速さ”と“アクセス性”)
- RNAscopeは1日で染色可能で、従来isHCR(全載3日、切片2日)は時間面で不利
- EC-isHCRはRNAscope並みの所要時間に到達し、この弱点を解消した
- さらに、RNAscopeはメーカーがプローブ配列を非公開である一方、著者らの枠組みは設計ツールを含めてオープンでアクセスしやすい
→ 速さだけでなく「研究者が自力で設計・拡張できる」点を価値として強調している。
smFISHとの比較(主に“サブセルラー解析”と“低コピーRNA”)
- smFISHは増幅なしのため、低コピーRNAではシグナルが控えめになり得る
- HCRは反応が急速に頭打ちになりにくく、反応時間を延ばすことでS/Nや低コピー検出を改善できるという性質がある
- そのうえで、EC-isHCRはサブセルラー局在(生殖顆粒、細胞突起)も示しており、サブセルラーRNA局在解析の実用的選択肢になり得る
→ 「速いHCR」であることが、smFISH領域にも食い込む可能性を示している。
6. 今後のインパクト:非モデル生物・多分野への波及
最後に著者らは、EC-isHCRの強みは非モデル生物にも広げやすい点にあると述べている。前提として、
- プローブ設計にはトランスクリプトーム(できればゲノム)が必要
- しかし近年、非モデル動物でもトランスクリプトーム・ゲノムデータが急速に蓄積している
その結果として、実際に本研究では(データ資源がある)D. sechellia、ニジマス、寄生蜂、アブラムシに適用でき、さらに寄生蜂・アブラムシへのisHCR適用は初報だ、と位置づけている。ここから、生態・進化の基礎研究だけでなく、医学・農学など応用分野にも広く貢献し得る、と結論づけている。
