Cell Resource Volume 187, Issue 23 p6760-6779.e24 November 14, 2024
Spatially exploring RNA biology in archival formalin-fixed paraffin-embedded tissues (アーカイブされたホルマリン固定パラフィン包埋組織におけるRNA生物学の空間的探求)
Zhiliang Bai 1, Dingyao Zhang 2, Yan Gao 3, Bo Tao 4, Daiwei Zhang 5, Shuozhen Bao 6, Archibald Enninful 6, Yadong Wang 7, Haikuo Li 6, Graham Su 6, Xiaolong Tian 6, Ningning Zhang 7, Yang Xiao 8, Yang Liu 4, Mark Gerstein 9, Mingyao Li 10, Yi Xing 11, Jun Lu 12, Mina L Xu 13, Rong Fan 14
Department of Biomedical Engineering, Yale University, New Haven, CT, USA (イェール大学)
Abstract
The capability to spatially explore RNA biology in formalin-fixed paraffin-embedded (FFPE) tissues holds transformative potential for histopathology research. Here, we present pathology-compatible deterministic barcoding in tissue (Patho-DBiT) by combining in situ polyadenylation and computational innovation for spatial whole transcriptome sequencing, tailored to probe the diverse RNA species in clinically archived FFPE samples. It permits spatial co-profiling of gene expression and RNA processing, unveiling region-specific splicing isoforms, and high-sensitivity transcriptomic mapping of clinical tumor FFPE tissues stored for 5 years. Furthermore, genome-wide single-nucleotide RNA variants can be captured to distinguish malignant subclones from non-malignant cells in human lymphomas. Patho-DBiT also maps microRNA regulatory networks and RNA splicing dynamics, decoding their roles in spatial tumorigenesis. Single-cell level Patho-DBiT dissects the spatiotemporal cellular dynamics driving tumor clonal architecture and progression. Patho-DBiT stands poised as a valuable platform to unravel rich RNA biology in FFPE tissues to aid in clinical pathology evaluation.
ホルマリン固定パラフィン包埋(FFPE)組織におけるRNA生物学の空間的探索能力は、組織病理学の研究に変革をもたらす可能性を秘めている。ここでは、臨床的に保存されたFFPEサンプル中の多様なRNA種をプローブするために調整された、空間的トランスクリプトームシーケンスのためのin situ ポリアデニレーションと計算機イノベーションを組み合わせた、組織における病理学的に適合する決定論的バーコード化(Patho-DBiT)を紹介する。これにより、遺伝子発現とRNAプロセシングの空間的共プロファイリングが可能になり、領域特異的スプライシングアイソフォームが明らかになり、5年間保存された臨床腫瘍FFPE組織の高感度トランスクリプトームマッピングが可能になる。さらに、ヒトリンパ腫における悪性サブクローンと非悪性細胞を区別するために、ゲノム全体の単一ヌクレオチドRNA変異を捕捉することができる。Patho-DBiTはまた、マイクロRNAの制御ネットワークやRNAスプライシングの動態をマッピングし、空間的腫瘍形成におけるそれらの役割を解読する。単一細胞レベルのPatho-DBiTは、腫瘍のクローン構造と進行を促す時空間的な細胞動態を解明する。Patho-DBiTは、FFPE組織における豊富なRNA生物学を解明し、臨床病理評価を支援する貴重なプラットフォームとなるであろう。
Keywords: RNA biology; clinical FFPE tissue; histopathology; microRNA; single-nucleotide RNA variants; spatial omics; spatiotemporal dynamics; splicing isoforms; whole transcriptome.
要約
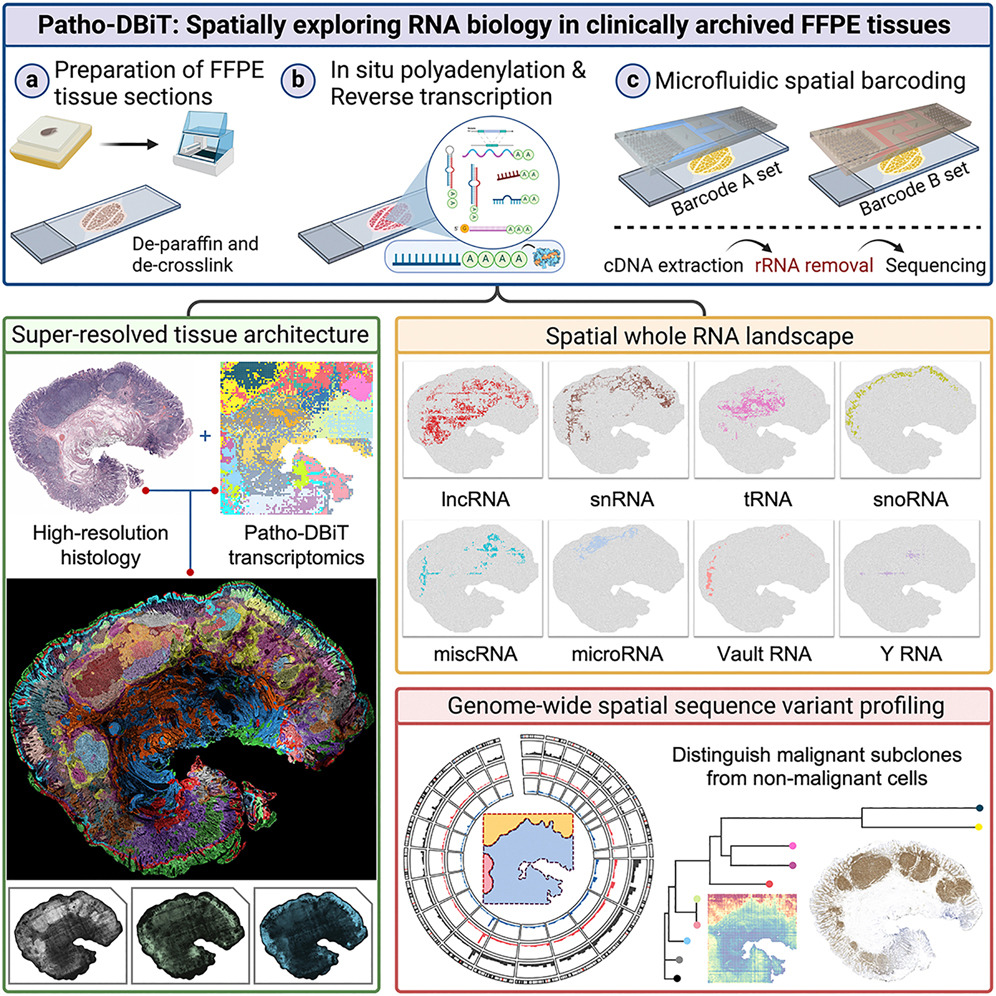
Patho-DBiT(Pathology-Compatible Deterministic Barcoding in Tissue)は、臨床で保存されているホルマリン固定パラフィン包埋(FFPE)組織でRNA種(mRNA、マイクロRNA、スプライシングアイソフォームなど)を空間的にプロファイリングする新技術である。この技術は腫瘍進展の空間的クローナルアーキテクチャを解析し、診断や治療戦略設計に新たな知見を提供する。この手法はRNA断片化や保存に伴う化学修飾の問題を克服し、多様なRNA種の解析を可能にするだけでなく、腫瘍形成や進展を細胞レベルで解明できるツールとして期待されている。
方法
- 組織内ポリアデニル化
RNA断片化を活用して、FFPE試料のRNA分子にポリアデニル化を付加した。この操作により、ポリAテールが欠損したRNA種(マイクロRNAや非コーディングRNAを含む)も解析可能にした。 - 空間バーコーディング
マイクロ流体デバイスを使用して組織にバーコードを付加した。このデバイスは50μmまたは100μmのチャネルを持ち、それぞれの位置情報を保持しながらRNA分子を空間的に区別する。これにより、各領域のRNAプロファイルを詳細に特定した。 - 組織処理とcDNA合成
試料は脱パラフィン化し、加熱による架橋解消を行った後、組織透過化を実施した。次に、酵素を用いてポリアデニル化を実施し、逆転写によってRNAからcDNAを合成した。これにより、断片化されたRNAでも効率的に解析可能となった。 - cDNAの抽出とライブラリー構築
cDNAを組織から抽出し、リボソームRNA(rRNA)の除去を行った。その後、テンプレートスイッチング法を用いて、バーコードを保持したまま増幅を行い、次世代シークエンシング用ライブラリーを作成した。 - 非コーディングRNAの解析
マイクロRNA、lncRNA、tRNA、snRNAなどの非コーディングRNAを対象に、広範なマッピング解析を実施した。これにより、従来技術では検出困難だった低豊富度RNAや非コーディングRNAの空間分布を明確化した。 - データ解析と統合
得られたRNA配列データをもとに、遺伝子発現マトリックスを生成し、非監督型クラスタリングを適用した。また、データセットをシングルセルRNAシークエンシング(scRNA-seq)参照データと統合し、細胞型や領域ごとのRNAプロファイルを精密に解析した。 - RNA編集とスプライシング解析
空間的RNA編集(A-to-I編集)やスプライシングアイソフォームの分布を解析し、各領域ごとのRNA加工プロファイルを特定した。特に、スプライシングイベントはPoly(A)テールによるRNA断片化補完によって、広範なカバレッジを達成した。 - SNVおよびCNV解析
RNAシークエンスデータを用いて単一ヌクレオチド変異(SNV)とコピー数変異(CNV)の分布を解析した。SNVは空間的な変異プロファイルを生成し、腫瘍内のクローナル構造を特定した。一方、CNV解析では、腫瘍進展や遺伝子領域のコピー数増加を詳細に評価した。 - 画像データとの統合
H&E染色や免疫組織化学(IHC)、in situハイブリダイゼーション(ISH)を行い、得られた空間的RNAデータと組織画像を統合した。これにより、遺伝子発現パターンと組織形態の関連性を高い解像度で明確化した。
この方法により、保存期間の長いFFPE試料から高感度なRNA生物学データを得ることができ、腫瘍進展や微小環境の空間的プロファイルを詳細に解析することが可能となった。
要約
Patho-DBiT(Pathology-Compatible Deterministic Barcoding in Tissue)は、臨床で保存されているホルマリン固定パラフィン包埋(FFPE)組織でRNA種(mRNA、マイクロRNA、スプライシングアイソフォームなど)を空間的にプロファイリングする新技術である。この技術は腫瘍進展の空間的クローナルアーキテクチャを解析し、診断や治療戦略設計に新たな知見を提供する。この手法はRNA断片化や保存に伴う化学修飾の問題を克服し、多様なRNA種の解析を可能にするだけでなく、腫瘍形成や進展を細胞レベルで解明できるツールとして期待されている。
結果
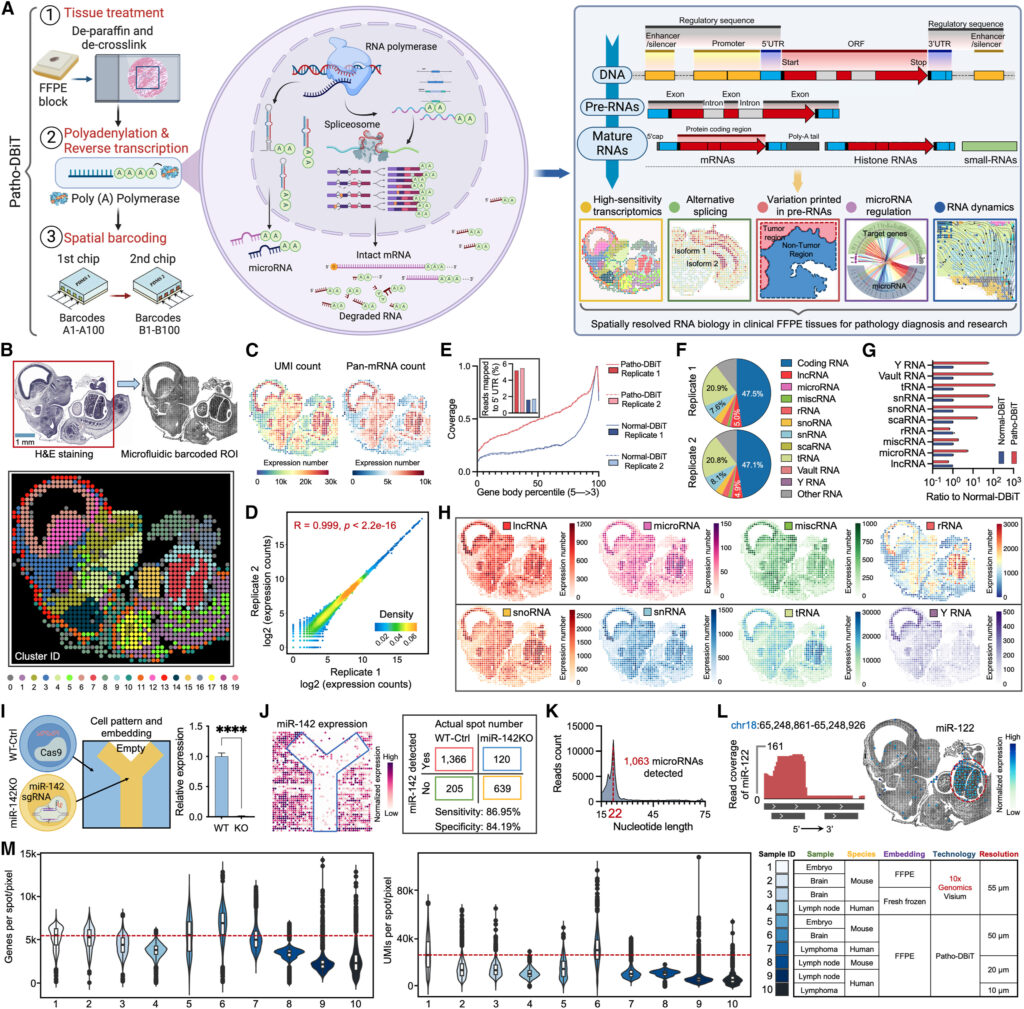
- RNA検出性能
Patho-DBiTでは、FFPE試料からピクセルごとに平均5,480の遺伝子と15,381のユニーク分子識別子(UMI)を検出した。非コーディングRNA(ncRNA:tRNA、snRNA、lncRNAなど)の捕捉率は既存技術を大幅に上回り、低豊富度RNA種の検出も実現した。また、リボソームRNA(rRNA)の除去率を高め、検出感度を向上させた。 - 腫瘍空間プロファイリング
AITL(アンジオ免疫芽球性T細胞リンパ腫)の5年間保存されたFFPE試料では、腫瘍領域のB細胞、T細胞、マクロファージの分布とそれらの遺伝子発現を空間的にマッピングした。また、マウス脳切片ではRNA A-to-I編集比率を地域ごとに詳細に解析し、視床で27.9%、繊維束で12.7%という明確な差を観察した。 - マイクロRNA解析
Patho-DBiTは2,300種類のヒト成熟マイクロRNAのうち1,352種類を検出した。腫瘍B細胞領域ではmiR-142、miR-146a、miR-150が高度に発現しており、これらが腫瘍形成に関与する可能性が示された。 - 腫瘍進展の解析
低悪性度MALTリンパ腫と高悪性度DLBCL(びまん性大細胞型B細胞リンパ腫)を比較した結果、DLBCLではNF-kBやPI3K/AKT経路が顕著に活性化し、p53やPTENといった抑制経路が低下していた。これにより腫瘍進展の分子メカニズムを空間的に明確化した。 - 空間的クローンアーキテクチャ
Patho-DBiTを用いたSNV(単一ヌクレオチド変異)とCNV(コピー数変異)の解析では、腫瘍B細胞領域において重要な変異(TBL1XR1、MAML2など)が特定された。また、腫瘍領域では染色体2、6、16のコピー数増加が見られた。
議論
- Patho-DBiTの強み
Patho-DBiTは、FFPE試料のRNA断片化やポリアデニル化の喪失など従来の制約を克服した。特に低豊富度RNAや非コーディングRNAを高感度で検出する能力が、既存技術(VisiumやDBiT-seq)よりも優れている。また、マイクロRNAやスプライシングアイソフォーム、RNA編集など幅広いRNA種を空間的に解析できる点で革新的である。これにより、腫瘍の微小環境の分子プロファイルを正確に描写できるようになった。 - 腫瘍進展の理解
低悪性度MALTリンパ腫から高悪性度DLBCLへの進展過程で、腫瘍内およびその周辺の分子動態を明確に解析できた。特にNF-kB経路やPI3K/AKT経路の活性化、p53やPTENの抑制が進展に深く関与していることが示された。また、腫瘍細胞と免疫細胞(例:マクロファージ)の空間的相互作用も明確化され、腫瘍微小環境の役割をより深く理解する手助けとなった。 - 病理学と診断への応用
Patho-DBiTはH&E染色やIHC解析と統合することで、より正確な腫瘍診断や個別化治療に向けた新しい情報を提供できる。また、SNVとCNVの空間プロファイルを組み合わせることで、薬剤耐性の可能性や腫瘍内の進展メカニズムを明確にする手段を提供する。 - 限界
Patho-DBiTは単一細胞レベルの解像度を持たない点で限界があるが、これにはさらなる技術開発が必要である。また、スプライシングイベント解析や抗原受容体の空間的定量化については、補助的な技術の統合が必要である。 - 臨床的意義と将来展望
Patho-DBiTは保存期間の長いFFPE試料からも高精度なデータを取得可能であり、腫瘍進展、薬剤耐性、免疫逃避のメカニズム解析に役立つ。将来的には、薬剤反応性の予測や免疫細胞戦略のデザインなど、治療法のパーソナライズに寄与する可能性がある。
Patho-DBiTは、腫瘍生物学および臨床病理学における強力なツールとして、新しい診断および治療の可能性を開く技術であるといえる。

青白く光る でんしんばしらは
月svjetlost(スビェトロスト)をすいあげてゐる
はげしくすきとほる そらの澱んだ 交響楽の底を
宮沢賢治『春と修羅』より「月夜のでんしんばしら」