Nature Neuroscience volume 25, pages1149–1162 (2022) Article
Open Access Published: 11 August 2022
Nina M. Dräger, Sydney M. Sattler, Cindy Tzu-Ling Huang, Olivia M. Teter, Kun Leng, Sayed Hadi Hashemi, Jason Hong, Giovanni Aviles, Claire D. Clelland, Lihong Zhan, Joe C. Udeochu, Lay Kodama, Andrew B. Singleton, Mike A. Nalls, Justin Ichida, Michael E. Ward, Faraz Faghri, Li Gan & Martin Kampmann
Institute for Neurodegenerative Diseases, University of California, San Francisco, San Francisco, CA, USA. Department of Biochemistry and Biophysics, University of California, San Francisco, San Francisco, CA, USA
〇論文紹介:YKさん(体育専門学群)
Abstract
Microglia are emerging as key drivers of neurological diseases. However, we lack a systematic understanding of the underlying mechanisms. Here, we present a screening platform to systematically elucidate functional consequences of genetic perturbations in human induced pluripotent stem cell-derived microglia. We developed an efficient 8-day protocol for the generation of microglia-like cells based on the inducible expression of six transcription factors. We established inducible CRISPR interference and activation in this system and conducted three screens targeting the ‘druggable genome’. These screens uncovered genes controlling microglia survival, activation and phagocytosis, including neurodegeneration-associated genes. A screen with single-cell RNA sequencing as the readout revealed that these microglia adopt a spectrum of states mirroring those observed in human brains and identified regulators of these states. A disease-associated state characterized by osteopontin (SPP1) expression was selectively depleted by colony-stimulating factor-1 (CSF1R) inhibition. Thus, our platform can systematically uncover regulators of microglial states, enabling their functional characterization and therapeutic targeting.
ミクログリアは、神経疾患の重要なドライバーとして浮上している。しかし、その基礎となるメカニズムの系統的な理解は不十分である。本論文では、ヒト人工多能性幹細胞(iPS細胞)由来のミクログリアにおける遺伝子異常の機能的影響を系統的に解明するためのスクリーニングプラットフォームを紹介する。筆者らは、6つの転写因子の発現を誘導し、8日間でミクログリア様細胞を効率的に作製するプロトコルを開発した。この系で誘導可能なCRISPRi (干渉interference)とCRISPRa (活性化activation) を確立し、「薬になるゲノム ‘druggable genome’」を標的とした3つのスクリーニングを実施した。これらのスクリーニングにより、神経変性関連遺伝子を含むミクログリアの生存、活性化、貪食を制御する遺伝子が発見された。また、単一細胞RNAシーケンスを用いたスクリーニングにより、これらのミクログリアがヒトの脳で観察されるようなスペクトルの状態をとることが明らかになり、これらの状態の制御因子も同定された。オステオポンチン(SPP1)の発現を特徴とする疾患関連状態は、コロニー刺激因子-1(CSF1R)阻害により選択的に減少した。このように、我々のプラットフォームは、ミクログリア状態の制御因子を系統的に発見し、その機能的特性評価と治療標的化を可能にするものである。
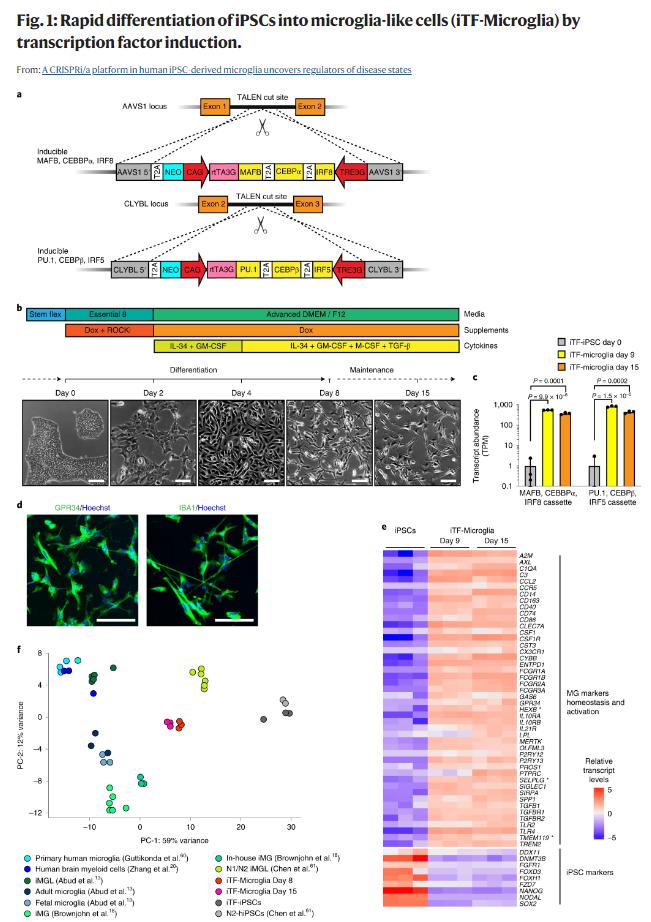
a, Strategy for stable integration of six transcription factors in AAVS1 and CLYBL loci by TALEN-mediated integration: The doxycycline-inducible reverse transcriptional activator (rtTA3G) is driven by the constitutive CAG promoter. Human MAFB, CEBPα and IRF8 are driven by the tet response element (TRE3G) in the AAVS1 locus. Human PU.1, CEBPβ and IRF5 are driven by TRE3G in the CLYBL locus. All transcription factors are separated from each other via T2A ribosome skipping sequences. b, Overview of the differentiation process for generating iTF-Microglia. Top, timeline with media, cytokines and doxycycline (Dox); bottom, representative phase-contrast images of cells on the indicated days. Scale bar, 100 μm. c, Expression of six inducible transcription factors during iTF-Microglia differentiation. Transcript abundance (transcripts per million, TPM) of MAFB, CEBPα, IRF8 cassette and the PU.1, CEBPβ, IRF5 cassette at day 0, day 9 and day 15 of differentiation. Mean ± s.d., n = 3 biological replicates, P values from two-tailed Student’s t-test. d, Representative immunofluorescence micrographs of iTF-Microglia on day 8 of differentiation stained for microglia markers GPR34 and IBA1. Nuclei were labeled by Hoechst 33342. Scale bar, 100 μm. e, Expression of iPSC and microglia marker genes in iPSCs and derived iTF-Microglia on day 9 and day 15 of differentiation. The heatmap displays normalized and gene-centered TPM counts for selected genes (rows) for three biological replicates of timepoints (columns). iTF-Microglia express microglia homeostatic markers and activation markers, while losing their expression of iPSC markers. Asterisks highlight microglia-selective markers. f, Principal component analysis (PCA) on the expression of microglia marker genes of iTF-Microglia, human adult ex vivo microglia60, fetal and adult microglia13, human myeloid cells20, other iPSC-microglia (iMG) / iPSC-microglia-like cells (iMGL)13,18,61 and iPSCs (this study and ref. 61). Each dot reflects an independent biological sample. Colors represent the different cell types.
a71f2d1497f7fae05c31c26632451a80YKさんは12月で当研究室のジャーナルクラブを卒業され、T大学大学院に進学されます。いつも洗練されたプレゼンテーションをしてくださいました。おめでとうございます!
